Published: 22 March 2023, MDPI
Deep Learning Approach to Classify Cutaneous Melanoma in a Whole Slide Image
Although the histopathological diagnosis of cutaneous melanocytic lesions is fairly accurate and reliable among experienced surgical pathologists, it is not perfect in every case (especially melanoma). Microscopic examination–clinicopathological correlation is the gold standard for the definitive diagnosis of melanoma. Pathologists may encounter diagnostic controversies when melanoma closely mimics Spitz’s nevus or blue nevus, exhibits amelanotic histopathology, or is in situ. It would be beneficial if diagnosing cutaneous melanocytic lesions can be automated by using deep learning, particularly when assisting surgical pathologists with their workloads. In this preliminary study, we investigated the application of deep learning for classifying cutaneous melanoma in whole-slide images (WSIs). We trained models via weakly supervised learning using a dataset of 66 WSIs (33 melanomas and 33 non-melanomas). We evaluated the models on a test set of 90 WSIs (40 melanomas and 50 non-melanomas), achieving ROC–AUC at 0.821 for the WSI level and 0.936 for the tile level by the best model. 論文詳細
Published: 05 January 2023, BioMed Central(BMC)
Inference of core needle biopsy whole slide images requiring definitive therapy for prostate cancer
【Background】
Prostate cancer is often a slowly progressive indolent disease. Unnecessary treatments from overdiagnosis are a significant concern, particularly low-grade disease. Active surveillance has being considered as a risk management strategy to avoid potential side effects by unnecessary radical treatment. In 2016, American Society of Clinical Oncology (ASCO) endorsed the Cancer Care Ontario (CCO) Clinical Practice Guideline on active surveillance for the management of localized prostate cancer.
【Methods】
Based on this guideline, we developed a deep learning model to classify prostate adenocarcinoma into indolent (applicable for active surveillance) and aggressive (necessary for definitive therapy) on core needle biopsy whole slide images (WSIs). In this study, we trained deep learning models using a combination of transfer, weakly supervised, and fully supervised learning approaches using a dataset of core needle biopsy WSIs (n=1300). In addition, we performed an inter-rater reliability evaluation on the WSI classification.
【Results】
We evaluated the models on a test set (n=645), achieving ROC-AUCs of 0.846 for indolent and 0.980 for aggressive. The inter-rater reliability evaluation showed s-scores in the range of 0.10 to 0.95, with the lowest being on the WSIs with both indolent and aggressive classification by the model, and the highest on benign WSIs.
【Conclusion】
The results demonstrate the promising potential of deployment in a practical prostate adenocarcinoma histopathological diagnostic workflow system. 論文詳細
Published: 4 December 2022, Proceedings of Machine Learning Research
Inference of captions from histopathological patches
Computational histopathology has made significant strides in the past few years, slowly getting closer to clinical adoption. One area of benefit would be the automatic generation of diagnostic reports from H&E-stained whole slide images which would further increase the efficiency of the pathologists’ routine diagnostic workflows. In this study, we compiled a dataset (PatchGastricADC22) of histopathological captions of stomach adenocarcinoma endoscopic biopsy specimens, which we extracted from diagnostic reports and paired with patches extracted from the associated whole slide images. The dataset contains a variety of gastric adenocarcinoma subtypes. We trained a baseline attention-based model to predict the captions from features extracted from the patches and obtained promising results. We make the captioned dataset of 262K patches publicly available. 論文詳細
Published: 30 December 2022, Cancers
Deep Learning-Based Screening of Urothelial Carcinoma in Whole Slide Images of Liquid-Based Cytology Urine Specimens
Urinary cytology is a useful, essential diagnostic method in routine urological clinical practice. Liquid-based cytology (LBC) for urothelial carcinoma screening is commonly used in the routine clinical cytodiagnosis because of its high cellular yields. Since conventional screening processes by cytoscreeners and cytopathologists using microscopes is limited in terms of human resources, it is important to integrate new deep learning methods that can automatically and rapidly diagnose a large amount of specimens without delay. The goal of this study was to investigate the use of deep learning models for the classification of urine LBC whole-slide images (WSIs) into neoplastic and non-neoplastic (negative). We trained deep learning models using 786 WSIs by transfer learning, fully supervised, and weakly supervised learning approaches. We evaluated the trained models on two test sets, one of which was representative of the clinical distribution of neoplastic cases, with a combined total of 750 WSIs, achieving an area under the curve for diagnosis in the range of 0.984–0.990 by the best model, demonstrating the promising potential use of our model for aiding urine cytodiagnostic processes. 論文詳細
Published: December 7, 2022, SAGE Publishing
Weakly Supervised Learning for Poorly Differentiated Adenocarcinoma Classification in Gastric Endoscopic Submucosal Dissection Whole Slide Images
Objective: Endoscopic submucosal dissection (ESD) is the preferred technique for treating early gastric cancers including poorly differentiated adenocarcinoma without ulcerative findings. The histopathological classification of poorly differentiated adenocarcinoma including signet ring cell carcinoma is of pivotal importance for determining further optimum cancer treatment(s) and clinical outcomes. Because conventional diagnosis by pathologists using microscopes is time-consuming and limited in terms of human resources, it is very important to develop computer-aided techniques that can rapidly and accurately inspect large number of histopathological specimen whole-slide images (WSIs). Computational pathology applications which can assist pathologists in detecting and classifying gastric poorly differentiated adenocarcinoma from ESD WSIs would be of great benefit for routine histopathological diagnostic workflow. Methods: In this study, we trained the deep learning model to classify poorly differentiated adenocarcinoma in ESD WSIs by transfer and weakly supervised learning approaches. Results: We evaluated the model on ESD, endoscopic biopsy, and surgical specimen WSI test sets, achieving and ROC-AUC up to 0.975 in gastric ESD test sets for poorly differentiated adenocarcinoma. Conclusion: The deep learning model developed in this study demonstrates the high promising potential of deployment in a routine practical gastric ESD histopathological diagnostic workflow as a computer-aided diagnosis system. 論文詳細
Published: November 23, 2022, PLOS
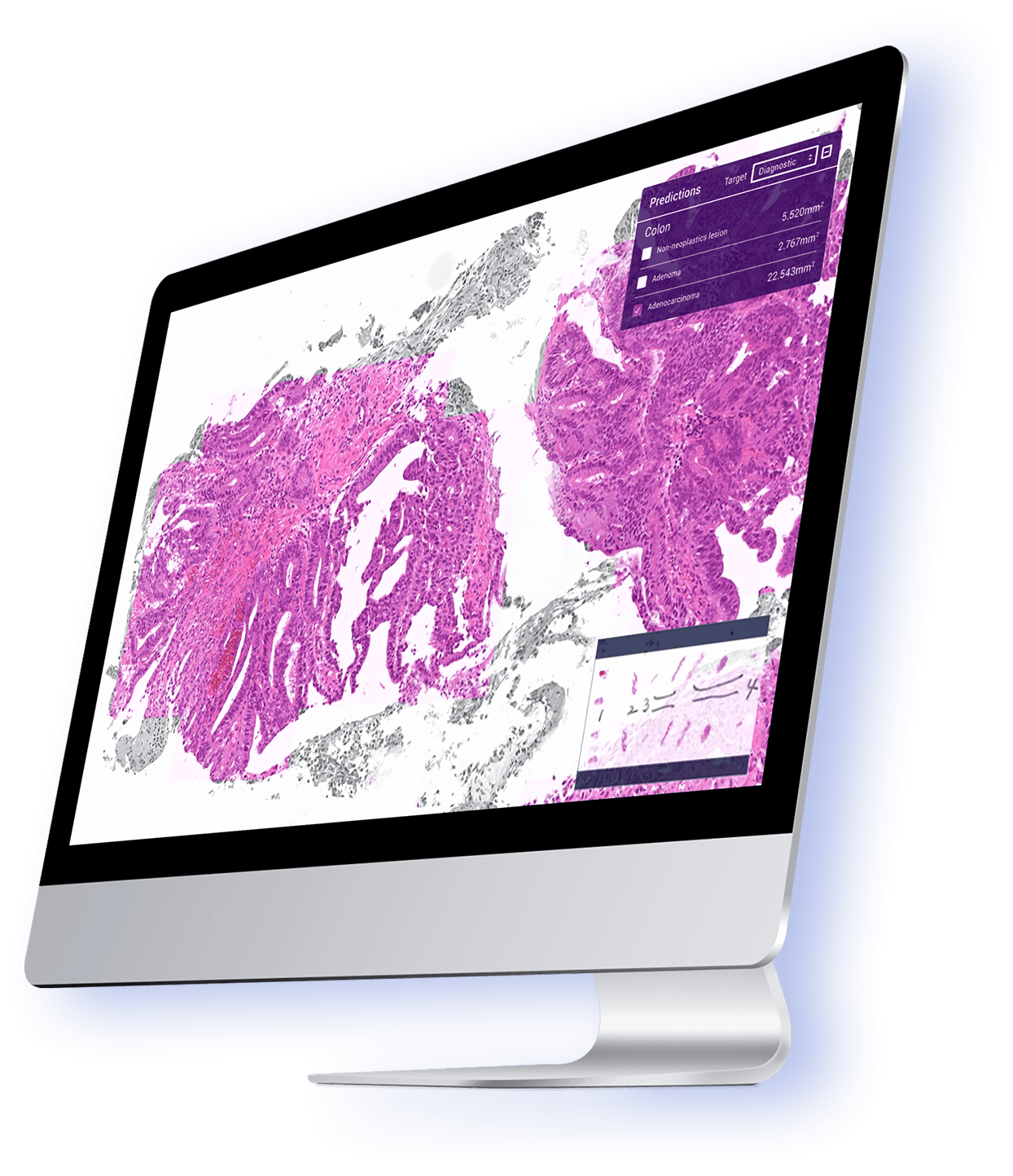
Weakly supervised learning for multi-organ adenocarcinoma classification in whole slide images
The primary screening by automated computational pathology algorithms of the presence or absence of adenocarcinoma in biopsy specimens (e.g., endoscopic biopsy, transbronchial lung biopsy, and needle biopsy) of possible primary organs (e.g., stomach, colon, lung, and breast) and radical lymph node dissection specimen is very useful and should be a powerful tool to assist surgical pathologists in routine histopathological diagnostic workflow. In this paper, we trained multi-organ deep learning models to classify adenocarcinoma in biopsy and radical lymph node dissection specimens whole slide images (WSIs). We evaluated the models on five independent test sets (stomach, colon, lung, breast, lymph nodes) to demonstrate the feasibility in multi-organ and lymph nodes specimens from different medical institutions, achieving receiver operating characteristic areas under the curves (ROC-AUCs) in the range of 0.91 -0.98. 論文詳細
Published: 28 September 2022, MDPI
Transfer Learning for Adenocarcinoma Classifications in the Transurethral Resection of Prostate Whole-Slide Images
The transurethral resection of the prostate (TUR-P) is an option for benign prostatic diseases, especially nodular hyperplasia patients who have moderate to severe urinary problems that have not responded to medication. Importantly, incidental prostate cancer is diagnosed at the time of TUR-P for benign prostatic disease. TUR-P specimens contain a large number of fragmented prostate tissues; this makes them time consuming to examine for pathologists as they have to check each fragment one by one. In this study, we trained deep learning models to classify TUR-P WSIs into prostate adenocarcinoma and benign (non-neoplastic) lesions using transfer and weakly supervised learning. We evaluated the models on TUR-P, needle biopsy, and The Cancer Genome Atlas (TCGA) public dataset test sets, achieving an ROC-AUC up to 0.984 in TUR-P test sets for adenocarcinoma. The results demonstrate the promising potential of deployment in a practical TUR-P histopathological diagnostic workflow system to improve the efficiency of pathologists. 論文詳細
Published: 21 March 2022, MDPI
A Deep Learning Model for Prostate Adenocarcinoma Classification in Needle Biopsy Whole-Slide Images Using Transfer Learning
The histopathological diagnosis of prostate adenocarcinoma in needle biopsy specimens is of pivotal importance for determining optimum prostate cancer treatment. Since diagnosing a large number of cases containing 12 core biopsy specimens by pathologists using a microscope is time-consuming manual system and limited in terms of human resources, it is necessary to develop new techniques that can rapidly and accurately screen large numbers of histopathological prostate needle biopsy specimens. Computational pathology applications that can assist pathologists in detecting and classifying prostate adenocarcinoma from whole-slide images (WSIs) would be of great benefit for routine pathological practice. In this paper, we trained deep learning models capable of classifying needle biopsy WSIs into adenocarcinoma and benign (non-neoplastic) lesions. We evaluated the models on needle biopsy, transurethral resection of the prostate (TUR-P), and The Cancer Genome Atlas (TCGA) public dataset test sets, achieving an ROC-AUC up to 0.978 in needle biopsy test sets and up to 0.9873 in TCGA test sets for adenocarcinoma. 論文詳細
Published: 24 February 2022, MDPI (Multidisciplinary Digital Publishing Institute)
A deep learning model for cervical cancer screening on liquid-based cytology specimens in whole slide images
Liquid-based cytology (LBC) for cervical cancer screening is now more common than the conventional smears, which when digitised from glass slides into whole-slide images (WSIs), opens up the possibility of artificial intelligence (AI)-based automated image analysis. Since conventional screening processes by cytoscreeners and cytopathologists using microscopes is limited in terms of human resources, it is important to develop new computational techniques that can automatically and rapidly diagnose a large amount of specimens without delay, which would be of great benefit for clinical laboratories and hospitals. The goal of this study was to investigate the use of a deep learning model for the classification of WSIs of LBC specimens into neoplastic and non-neoplastic. To do so, we used a dataset of 1605 cervical WSIs. We evaluated the model on three test sets with a combined total of 1468 WSIs, achieving ROC AUCs for WSI diagnosis in the range of 0.89–0.96, demonstrating the promising potential use of such models for aiding screening processes. 論文詳細
Published: 25 January 2022, Springer Nature
A deep learning model for breast ductal carcinoma in situ classification in whole slide images
The pathological differential diagnosis between breast ductal carcinoma in situ (DCIS) and invasive ductal carcinoma (IDC) is of pivotal importance for determining optimum cancer treatment(s) and clinical outcomes. Since conventional diagnosis by pathologists using microscopes is limited in terms of human resources, it is necessary to develop new techniques that can rapidly and accurately diagnose large numbers of histopathological specimens. Computational pathology tools which can assist pathologists in detecting and classifying DCIS and IDC from whole slide images (WSIs) would be of great benefit for routine pathological diagnosis. In this paper, we trained deep learning models capable of classifying biopsy and surgical histopathological WSIs into DCIS, IDC, and benign. We evaluated the models on two independent test sets (n= 1382, n= 548), achieving ROC areas under the curves (AUCs) up to 0.960 and 0.977 for DCIS and IDC, respectively. 論文詳細
Published: 9 November 2021, MDPI (Multidisciplinary Digital Publishing Institute)
Deep learning models for poorly differentiated colorectal adenocarcinoma classification in whole slide images using transfer learning
Colorectal poorly differentiated adenocarcinoma (ADC) is known to have a poor prognosis as compared with well to moderately differentiated ADC. The frequency of poorly differentiated ADC is relatively low (usually less than 5% among colorectal carcinomas). Histopathological diagnosis based on endoscopic biopsy specimens is currently the most cost effective method to perform as part of colonoscopic screening in average risk patients, and it is an area that could benefit from AI-based tools to aid pathologists in their clinical workflows. In this study, we trained deep learning models to classify poorly differentiated colorectal ADC from Whole Slide Images (WSIs) using a simple transfer learning method. We evaluated the models on a combination of test sets obtained from five distinct sources, achieving receiver operating characteristic curve (ROC) area under the curves (AUCs) up to 0.95 on 1799 test cases. 論文詳細
Published: 26 October 2021, MDPI (Multidisciplinary Digital Publishing Institute)
Breast invasive ductal carcinoma classification on whole slide images with weakly-supervised and transfer learning
Invasive ductal carcinoma (IDC) is the most common form of breast cancer. For the non-operative diagnosis of breast carcinoma, core needle biopsy has been widely used in recent years for the evaluation of histopathological features, as it can provide a definitive diagnosis between IDC and benign lesion (e.g., fibroadenoma), and it is cost effective. Due to its widespread use, it could potentially benefit from the use of AI-based tools to aid pathologists in their pathological diagnosis workflows. In this paper, we trained invasive ductal carcinoma (IDC) whole slide image (WSI) classification models using transfer learning and weakly-supervised learning. We evaluated the models on a core needle biopsy (n = 522) test set as well as three surgical test sets (n = 1129) obtaining ROC AUCs in the range of 0.95–0.98. The promising results demonstrate the potential of applying such models as diagnostic aid tools for pathologists in clinical practice. 論文詳細
Published: 14 October 2021, Scientific Reports
A deep learning model for gastric diffuse-type adenocarcinoma classification in whole slide images
Gastric diffuse-type adenocarcinoma represents a disproportionately high percentage of cases of gastric cancers occurring in the young, and its relative incidence seems to be on the rise. Usually it affects the body of the stomach, and it presents shorter duration and worse prognosis compared with the differentiated (intestinal) type adenocarcinoma. The main difficulty encountered in the differential diagnosis of gastric adenocarcinomas occurs with the diffuse-type. As the cancer cells of diffuse-type adenocarcinoma are often single and inconspicuous in a background desmoplaia and inflammation, it can often be mistaken for a wide variety of non-neoplastic lesions including gastritis or reactive endothelial cells seen in granulation tissue. In this study we trained deep learning models to classify gastric diffuse-type adenocarcinoma from WSIs. We evaluated the models on five test sets obtained from distinct sources, achieving receiver operator curve (ROC) area under the curves (AUCs) in the range of 0.95–0.99. The highly promising results demonstrate the potential of AI-based computational pathology for aiding pathologists in their diagnostic workflow system. 論文詳細
Modified: 27 Aug 2021, Proceedings of Machine Learning Research
Partial transfusion: on the expressive influence of trainable batch norm parameters for transfer learning
Transfer learning from ImageNet is the go-to approach when applying deep learning to medical images. The approach is either to fine-tune a pre-trained model or use it as a feature extractor. Most modern architecture contain batch normalisation layers, and fine-tuning a model with such layers requires taking a few precautions as they consist of trainable and non-trainable weights and have two operating modes: training and inference. Attention is primarily given to the non-trainable weights used during inference, as they are the primary source of unexpected behaviour or degradation in performance during transfer learning. It is typically recommended to fine-tune the model with the batch normalisation layers kept in inference mode during both training and inference. In this paper, we pay closer attention instead to the trainable weights of the batch normalisation layers, and we explore their expressive influence in the context of transfer learning. We find that only fine-tuning the trainable weights (scale and centre) of the batch normalisation layers leads to similar performance as to fine-tuning all of the weights, with the added benefit of faster convergence. We demonstrate this on a variety of seven publicly available medical imaging datasets, using four different model architectures. 論文詳細
Published: 08 Feb 2021 (modified: 21 Apr 2021), OpenReview.net
Partial transfusion: on the expressive influence of trainable batch norm parameters for transfer learning
Transfer learning from ImageNet is the go-to approach when applying deep learning to medical images. The approach is either to fine-tune a pre-trained model or use it as a feature extractor.
Most modern architecture contain batch normalisation layers, and fine-tuning a model with such layers requires taking a few precautions as they consist of trainable and non-trainable weights and have two operating modes: training and inference. Attention is primarily given to the non-trainable weights used during inference, as they are the primary source of unexpected behaviour or degradation in performance during transfer learning. It is typically recommended to fine-tune the model with the batch normalisation layers kept in inference mode during both training and inference.
In this paper, we pay closer attention instead to the trainable weights of the batch normalisation layers, and we explore their expressive influence in the context of transfer learning.
We find that only fine-tuning the trainable weights (scale and centre) of the batch normalisation layers leads to similar performance as to fine-tuning all of the weights, with the added benefit of faster convergence. We demonstrate this on a variety of seven publicly available medical imaging datasets, using four different model architectures. 論文詳細
Published: 30 June 2021, Technology in Cancer Research & Treatment
Deep Learning Models for Gastric Signet Ring Cell Carcinoma Classification in Whole Slide Images
Signet ring cell carcinoma (SRCC) of the stomach is a rare type of cancer with a slowly rising incidence. It tends to be more difficult to detect by pathologists, mainly due to its cellular morphology and diffuse invasion manner, and it has poor prognosis when detected at an advanced stage. Computational pathology tools that can assist pathologists in detecting SRCC would be of a massive benefit. In this paper, we trained deep learning models using transfer learning, fully-supervised learning, and weakly-supervised learning to predict SRCC in Whole Slide Images (WSIs) using a training set of 1,765 WSIs. We evaluated the models on two different test sets (n = 999, n = 455). The best model achieved a ROC-AUC of at least 0.99 on all two test sets, setting a top baseline performance for SRCC WSI classification. 論文詳細
Published: 19 April 2021, Scientific Reports
A deep learning model to detect pancreatic ductal adenocarcinoma on endoscopic ultrasound-guided fine-needle biopsy
Histopathological diagnosis of pancreatic ductal adenocarcinoma (PDAC) on endoscopic ultrasonography-guided fine-needle biopsy (EUS-FNB) specimens has become the mainstay of preoperative pathological diagnosis. However, on EUS-FNB specimens, accurate histopathological evaluation is difficult due to low specimen volume with isolated cancer cells and high contamination of blood, inflammatory and digestive tract cells. In this study, we performed annotations for training sets by expert pancreatic pathologists and trained a deep learning model to assess PDAC on EUS-FNB of the pancreas in histopathological whole-slide images. We obtained a high receiver operator curve area under the curve of 0.984, accuracy of 0.9417, sensitivity of 0.9302 and specificity of 0.9706. Our model was able to accurately detect difficult cases of isolated and low volume cancer cells. If adopted as a supportive system in routine diagnosis of pancreatic EUS-FNB specimens, our model has the potential to aid pathologists diagnose difficult cases. 論文詳細
Published: 14 April 2021, Scientific Reports
A deep learning model for the classification of indeterminate lung carcinoma in biopsy whole slide images
The differentiation between major histological types of lung cancer, such as adenocarcinoma (ADC), squamous cell carcinoma (SCC), and small-cell lung cancer (SCLC) is of crucial importance for determining optimum cancer treatment. Hematoxylin and Eosin (H&E)-stained slides of small transbronchial lung biopsy (TBLB) are one of the primary sources for making a diagnosis; however, a subset of cases present a challenge for pathologists to diagnose from H&E-stained slides alone, and these either require further immunohistochemistry or are deferred to surgical resection for definitive diagnosis. We trained a deep learning model to classify H&E-stained Whole Slide Images of TBLB specimens into ADC, SCC, SCLC, and non-neoplastic using a training set of 579 WSIs. The trained model was capable of classifying an independent test set of 83 challenging indeterminate cases with a receiver operator curve area under the curve (AUC) of 0.99. We further evaluated the model on four independent test sets—one TBLB and three surgical, with combined total of 2407 WSIs—demonstrating highly promising results with AUCs ranging from 0.94 to 0.99. 論文詳細
Published: 09 June 2020, Scientific Reports
Weakly-supervised learning for lung carcinoma classification using deep learning
Lung cancer is one of the major causes of cancer-related deaths in many countries around the world, and its histopathological diagnosis is crucial for deciding on optimum treatment strategies. Recently, Artificial Intelligence (AI) deep learning models have been widely shown to be useful in various medical fields, particularly image and pathological diagnoses; however, AI models for the pathological diagnosis of pulmonary lesions that have been validated on large-scale test sets are yet to be seen. We trained a Convolution Neural Network (CNN) based on the EfficientNet-B3 architecture, using transfer learning and weakly-supervised learning, to predict carcinoma in Whole Slide Images (WSIs) using a training dataset of 3,554 WSIs. We obtained highly promising results for differentiating between lung carcinoma and non-neoplastic with high Receiver Operator Curve (ROC) area under the curves (AUCs) on four independent test sets (ROC AUCs of 0.975, 0.974, 0.988, and 0.981, respectively). Development and validation of algorithms such as ours are important initial steps in the development of software suites that could be adopted in routine pathological practices and potentially help reduce the burden on pathologists. 論文詳細
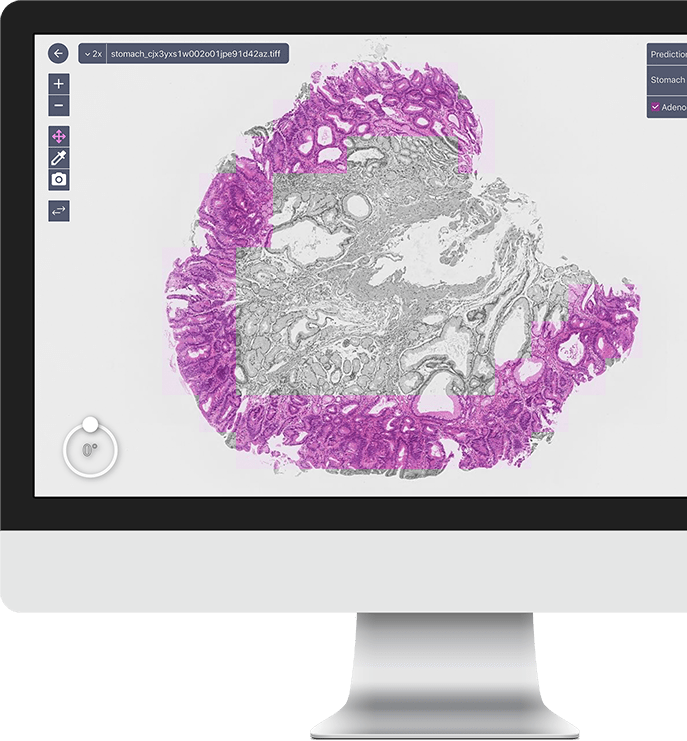
Published: 30 January 2020, Scientific Reports
Deep Learning Models for Histopathological Classification of Gastric and Colonic Epithelial Tumours
Histopathological classification of gastric and colonic epithelial tumours is one of the routine pathological diagnosis tasks for pathologists. Computational pathology techniques based on Artificial intelligence (AI) would be of high benefit in easing the ever increasing workloads on pathologists, especially in regions that have shortages in access to pathological diagnosis services. In this study, we trained convolutional neural networks (CNNs) and recurrent neural networks (RNNs) on biopsy histopathology whole-slide images (WSIs) of stomach and colon. The models were trained to classify WSI into adenocarcinoma, adenoma, and non-neoplastic. We evaluated our models on three independent test sets each, achieving area under the curves (AUCs) up to 0.97 and 0.99 for gastric adenocarcinoma and adenoma, respectively, and 0.96 and 0.99 for colonic adenocarcinoma and adenoma respectively. The results demonstrate the generalisation ability of our models and the high promising potential of deployment in a practical histopathological diagnostic workflow system. 論文詳細